Déploiement de modèles ML/DL dans une pile de services de démonstration d'IA consolidée
Keywords: IRIS, IntegratedML, Flask, FastAPI, Tensorflow servant, HAProxy, Docker, Covid-19
Objective:
Nous avons abordé quelques démonstrations rapides d'apprentissage profond et d'apprentissage automatique au cours des derniers mois, notamment un simple classificateur d'images radiographiques Covid-19 et un classificateur de résultats de laboratoire Covid-19 pour les admissions possibles en soins intensifs. Nous avons également évoqué une implémentation de démonstration IntegratedML du classificateur ICU. Alors que la randonnée de la "science des données" se poursuit, le moment est peut-être venu d'essayer de déployer des services d'IA du point de vue de "l'ingénierie des données" - pourrions-nous regrouper tout ce que nous avons abordé jusqu'à présent dans un ensemble d'API de services ? Quels sont les outils, les composants et l'infrastructure communs que nous pourrions exploiter pour réaliser une telle pile de services dans son approche la plus simple possible ?
Cadre
Dans le cadre de ce qui suit:
Pour commencer, nous pouvons simplement utiliser docker-compose pour déployer les composants dockerisés suivants dans un serveur AWS Ubuntu
- HAProxy - équilibreur de charge
- Gunicorn vs. Univorn ** - passerelle web **serveurs
- Flask vs. FastAPI - serveurs d'application pour l'interface utilisateur des applications Web, les définitions des API de service, la génération des cartes thermiques, etc.
- Tensorflow Model Serving vs. Tensorflow-GPU Model Serving - serveurs d'applications pour les classifications d'images, etc.
- IRIS IntegratedML - AutoML consolidé App+DB avec interface SQL.
- Python3 dans Jupyter Notebook pour émuler un client pour le benchmarking.
- Docker et docker-compose.
- AWS Ubuntu 16.04 avec un GPU Tesla T4
Remarque: Tensorflow Serving avec GPU n'est utilisé qu'à des fins de démonstration - vous pouvez simplement désactiver l'image liée au GPU (dans un fichier docker) et la configuration (dans le fichier docker-compose.yml).
Out of scope (Hors de portée) ou sur la prochaine liste de souhaits :
- **Les serveurs web Nginx ou Apache etc. sont omis dans la démo pour le moment.
- RabbitMQ et Redis - courtier de file d'attente pour une messagerie fiable qui peut être remplacé par IRIS ou Ensemble.
IAM (Intersystems API Manger) ou Kong est sur la liste des souhaits. - SAM(Intersystems System Alert & Monitoring)
- ICM (Intersystems Cloud Manager) avec l'opérateur Kubernetes - toujours l'un de mes préférés depuis sa naissance
- FHIR (serveur FHIR R4 basé sur Intesystems IRIS et FHIR Sandbox pour les applications SMART sur FHIR)
- Outils de développement CI/CD ou Github Actions.
De toute façon, un "ingénieur en apprentissage automatique" ("Machine Learning Engineer") mettra inévitablement la main sur ces composants pour fournir des environnements de production tout au long des cycles de vie des services. Nous pourrons en savoir plus au fil du temps.
Dépôt Github
Le code source complet se trouve à l'adresse suivante : https://github.com/zhongli1990/covid-ai-demo-deployment
Le référentiel integratedML-demo-template est également réutilisé avec le nouveau référentiel.
Modèle de déploiement
Le schéma de déploiement logique de ce cadre de test "Démonstration de l'IA dans les Dockers" est présenté ci-dessous.
.png)
Pour la démonstration, j'ai délibérément créé deux piles distinctes pour la classification de l'apprentissage profond et le rendu web, puis j'ai utilisé un HAProxy comme équilibreur de charge pour distribuer les requêtes API entrantes entre ces deux piles de manière indépendante.
- Guniorn + Flask + Tensorflow Serving
- Univcorn + FaskAPI + Tensorflow Serving GPU
IRIS avec IntegratedML est utilisé pour les échantillons de démonstration d'apprentissage automatique, comme dans l'article précédent de prédiction de l'ICU.
J'ai omis certains composants communs dans la démo actuelle qui seraient nécessaires ou envisagés pour les services de production :
- Serveurs Web : Nginx ou Apache, etc. Ils seront nécessaires entre HAProxy et Gunicorn/Uvicorn, pour une gestion correcte des sessions HTTP, c'est-à-dire pour éviter les attaques DoS, etc.
- Gestionnaire de file d'attente et bases de données : RabbitMQ et/ou Redis, etc., entre Flask/FastAPI et le serveur backend, pour un service asynchrone fiable et la persistance des données/configurations, etc.
- Passerelle API : IAM ou Kong clusters, entre l'équilibreur de charge HAProxy et le serveur web pour la gestion des API sans créer de point singulier de défaillance.
- Surveillance et alerte : SAM serait bien.
- Provisionnement pour CI/CD devops : ICM avec K8s serait nécessaire pour le déploiement et la gestion neutre en nuage, et pour CI/CD avec d'autres outils devops communs.
En fait, IRIS lui-même peut certainement être utilisé comme gestionnaire de file d'attente de niveau entreprise ainsi que comme base de données performante pour une messagerie fiable. Dans l'analyse des modèles, il apparaît qu'IRIS peut remplacer les courtiers de file d'attente et les bases de données RabbitMQ/Redis/MongoDB, etc., et qu'il serait donc mieux consolidé avec une latence bien moindre et de meilleures performances globales. Et plus encore, IRIS Web Gateway (anciennement CSP Gateway) peut certainement être positionné à la place de Gunicorn ou Unicorn, etc, n'est-ce pas ?
Topologie de l'environnement
Il existe quelques options courantes pour mettre en œuvre le modèle logique ci-dessus dans tous les composants Docker. Les plus courantes sont les suivantes :
- docker-compose
- docker swarm etc
- Kubernetes etc
- ICM avec K8s Operation
Cette démonstration commence avec "docker-compose" pour un PoC fonctionnel et un certain benchmarking. Nous aimerions certainement utiliser K8s et peut-être aussi ICM au fil du temps.
Comme décrit dans son fichier docker-compose.yml, une implémentation physique de sa topologie d'environnement sur un serveur AWS Ubuntu ressemblerait à ceci :
.png)
Le diagramme ci-dessus montre comment ces ports de service de toutes les instances Docker sont mappés et exposés directement sur le serveur Ubuntu à des fins de démonstration. En production, la sécurité devrait être renforcée. Et pour les besoins de la démonstration, tous les conteneurs sont connectés au même réseau Docker, alors qu'en production, il serait séparé en routable externe et non-routable interne.
Composants "Dockerisés"
Le tableau ci-dessous montre comment les volumes de stockage de la machine hôte sont montés sur chaque instance de conteneur comme spécifié dans ce fichier docker-compose.yml :
ubuntu@ip-172-31-35-104:/zhong/flask-xray$ tree ./ -L 2
./├── covid19 (Les conteneurs Flask+Gunicorn et Tensorflow Serving seront montés ici)│├── app.py (Flask main app: Les interfaces de l'application web et du service API sont définies et mises en œuvre ici)│├── covid19_models (Les modèles Tensorflow sont publiés et versionnés ici pour la classification des images Le conteneur Tensorflow Serving avec CPU)│├── Dockerfile (Le serveur Flask avec Gunicorn: CMD ["gunicorn", "app:app", "--bind", "0.0.0.0:5000", "--workers", "4", "--threads", "2"])│├── models (Modèles au format .h5 pour l'application Flask et démonstration API de la génération de heatmaps par grad-cam sur des radiographies.)│├── __pycache__│├── README.md│├── requirements.txt (Paquets Python nécessaires pour les applications complètes de Flask+Gunicorn) │├── scripts│├── static (Fichiers statiques Web)│├── templates (Modèles de rendu Web)│├── tensorflow_serving (Fichier de configuration pour le service de tensorflow)│└── test_images├── covid-fastapi (Les conteneurs FastAPI+Uvicorn et Tensorflow Serving avec GPU seront définis ici)│├── covid19_models (Les modèles Tensorflow au service des GPU sont publiés et versionnés ici pour la classification des images)│├── Dockerfile (Le serveur Uvicorn+FastAPI sera lancé ici: CMD ["uvicorn", "main:app", "--host", "0.0.0.0", "--port", "8000", "--workers", "4" ])│├── main.py (FastAPI app: les interfaces de l'application web et du service API sont définies et mises en œuvre ici)│├── models (Modèles au format .h5 pour l'application FastAPI et démonstration API de la génération de heatmaps par grad-cam sur des radiographies)│├── __pycache__│├── README.md│├── requirements.txt│├── scripts│├── static│├── templates│├── tensorflow_serving│└── test_images├── docker-compose.yml (Full stack Docker definition file. La version 2.3 est utilisée pour tenir compte du GPU Docker "nvidia runtime", sinon la version 3.x peut être utilisée)├── haproxy (Le service docker HAProxy est défini ici. Note : une session collante peut être définie pour le backend LB. ) │├── Dockerfile│└── haproxy.cfg└── notebooks (Le service conteneur Jupyter Notebook avec Tensorflow 2.2 et Tensorboard etc)├── Dockerfile├── notebooks (Exemples de fichiers notebook pour émuler des applications API Client externes pour les tests fonctionnels et les tests de référence API en Python sur l'équilibreur de charge, etc)└── requirements.txt
Remarque: Le docker-compose.yml ci-dessus est destiné à la démonstration d'apprentissage profond de Convid X-Rays. Il est utilisé avec le docker-compose.yml d'un autre integratedML-demo-template pour former la pile de services complète, comme indiqué dans la topologie de l'environnement.
Démarrage des services
Un simple docker-compose up -d permettrait de démarrer tous les services de conteneurs :
ubuntu@ip-172-31-35-104:~$ docker ps
ID DE CONTENEUR IMAGE COMMANDE STATUT CRÉÉ PORTS NOMS
31b682b6961d iris-aa-server:2020.3AA "/iris-main" Il y a 7 semaines Jusqu'à 2 jours (en bonne santé) 2188/tcp, 53773/tcp, 54773/tcp, 0.0.0.0:8091->51773/tcp, 0.0.0.0:8092->52773/tcp iml-template-master_irisimlsvr_1
6a0f22ad3ffc haproxy:0.0.1 "/docker-entrypoint.…" Il y a 8 semaines Jusqu'à 2 jours 0.0.0.0:8088->8088/tcp flask-xray_lb_1
71b5163d8960 ai-service-fastapi:0.2.0 "uvicorn main:app --…" Il y a 8 semaines Jusqu'à 2 jours 0.0.0.0:8056->8000/tcp flask-xray_fastapi_1
400e1d6c0f69 tensorflow/serving:latest-gpu "/usr/bin/tf_serving…" Il y a 8 semaines Jusqu'à 2 jours 0.0.0.0:8520->8500/tcp, 0.0.0.0:8521->8501/tcp flask-xray_tf2svg2_1
eaac88e9b1a7 ai-service-flask:0.1.0 "gunicorn app:app --…" Шl y a 8 semaines Jusqu'à 2 jours 0.0.0.0:8051->5000/tcp flask-xray_flask_1
e07ccd30a32b tensorflow/serving "/usr/bin/tf_serving…" Il y a 8 semaines Jusqu'à 2 jours 0.0.0.0:8510->8500/tcp, 0.0.0.0:8511->8501/tcp flask-xray_tf2svg1_1
390dc13023f2 tf2-jupyter:0.1.0 "/bin/sh -c '/bin/ba…" Il y a 8 semaines Jusqu'à 2 jours 0.0.0.0:8506->6006/tcp, 0.0.0.0:8586->8888/tcp flask-xray_tf2jpt_1
88e8709404ac tf2-jupyter-jdbc:1.0.0-iml-template "/bin/sh -c '/bin/ba…" Il y a 2 $ois Jusqu'à 2 jours 0.0.0.0:6026->6006/tcp, 0.0.0.0:8896->8888/tcp iml-template-master_tf2jupyter_1
docker-compose up --scale fastapi=2 --scale flask=2 -d par exemple, sera mis à l'échelle horizontalement jusqu'à 2 conteneurs Gunicorn+Flask et 2 conteneurs Univcorn+FastAPI :
ubuntu@ip-172-31-35-104:/zhong/flask-xray$ docker ps
ID DE CONTENEUR IMAGE COMMANDE STATUT CRÉÉ PORTS NOMS
dbee3c20ea95 ai-service-fastapi:0.2.0 "uvicorn main:app --…" Il y a 4 minutes Jusqu'à 4 minutes 0.0.0.0:8057->8000/tcp flask-xray_fastapi_2
95bcd8535aa6 ai-service-flask:0.1.0 "gunicorn app:app --…" Il y a 4 minutes Jusqu'à 4 minutes 0.0.0.0:8052->5000/tcp flask-xray_flask_2
... ...
L'exécution d'un autre "docker-compose up -d" dans le répertoire de travail de "integrtedML-demo-template" a fait apparaître le conteneur irisimlsvr et tf2jupyter dans la liste ci-dessus.
Tests
1. Application web de démonstration de l'IA avec une interface utilisateur simple
Après avoir démarré les services docker ci-dessus, nous pouvons visiter une application web de démonstration pour X-Ray Covid-19 lung detection hébergée dans une instance AWS EC2 à une adresse temporaire à http://ec2-18-134-16-118.eu-west-2.compute.amazonaws.com:8056/
Voici ci-dessous quelques écrans capturés depuis mon mobile. L'interface utilisateur de démonstration est très simple : en gros, je clique sur "Choose File" puis sur le bouton "Submit" pour télécharger une image radiographique, puis l'application affiche un rapport de classification. S'il s'agit d'une radiographie Covid-19, une [carte thermique sera affichée] (https://community.intersystems.com/post/explainability-and-visibility-covid-19-x-ray-classifiers-deep-learning) pour reproduire la zone de lésion "détectée" par DL ; sinon, le rapport de classification ne montrera que l'image radiographique téléchargée.
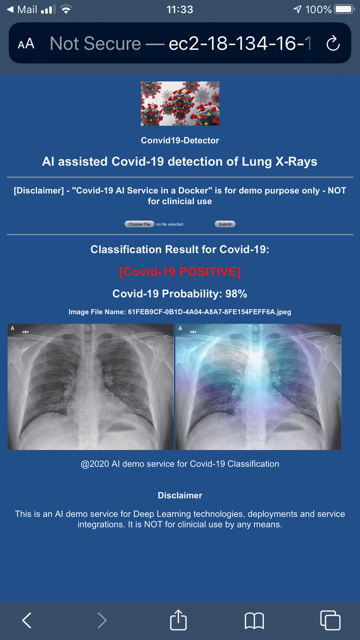
L'application web est une page serveur Python dont la logique est principalement codée dans le fichier main.py de FastAPI, ainsi que dans le fichier app.py de Flask.
Quand j'aurai un peu plus de temps libre, je pourrais documenter en détail les différences de codage et de convention entre Flask et FastAPI. En fait, j'espère pouvoir faire un hébergement de démonstration Flask vs FastAPI vs IRIS pour l'IA.
2. Test des API de démonstration
FastAPI (exposé au port 8056) a intégré des documents Swagger API, comme indiqué ci-dessous. C'est très pratique. Tout ce que j'ai à faire est d'utiliser "/docs" dans son URL, par exemple :
.png)
J'ai intégré quelques paramètres (tels que /hello et /items) et quelques interfaces API de démonstration (telles que /healthcheck, /predict, et predict/heatmap).
Testons rapidement ces API, en exécutant quelques lignes Python (en tant qu'émulateur d'application client API) dans l'un des [fichiers d'échantillons de Jupyter Notebook que j'ai créés] (https://github.com/zhongli1990/covid-ai-demo-deployment/tree/master/notebooks/notebooks) pour ce service de démonstration de l'IA.
Ci-dessous, j'exécute ce fichier à titre d'exemple : https://github.com/zhongli1990/covid-ai-demo-deployment/blob/master/notebooks/notebooks/Covid19-3class-Heatmap-Flask-FastAPI-TF-serving-all-in-one-HAProxy2.ipynb
Tout d'abord pour tester que le backend TF-Serving (port 8511) et TF-Serving-GPU (port 8521) sont en place et fonctionnent :
!curl http://172.17.0.1:8511/v1/models/covid19 # servant tensorflow
!curl http://172.17.0.1:8521/v1/models/covid19 # servant tensorflow-gpu{
"model_version_status": [
{
"version": "2",
"state": "AVAILABLE",
"status": {
"error_code": "OK",
"error_message": ""
}
}
]
}
{
"model_version_status": [
{
"version": "2",
"state": "AVAILABLE",
"status": {
"error_code": "OK",
"error_message": ""
}
}
]
}
Ensuite, vérifiez que les services API suivants sont en place et fonctionnent :
r = requests.get('http://172.17.0.1:8051/covid19/api/v1/healthcheck') # tf servant le docker avec le cpu
print(r.status_code, r.text)
r = requests.get('http://172.17.0.1:8056/covid19/api/v1/healthcheck') # tf-servant le docker avec le gpu
print(r.status_code, r.text)
r = requests.get('http://172.17.0.1:8088/covid19/api/v1/healthcheck') # tf-servant le docker avec le HAproxy
print(r.status_code, r.text)Et les résultats attendus seraient :
200 L'API du détecteur Covid19 est en ligne ! 200 "L'API du détecteur Covid19 est en ligne !\n\n" 200 "L'API du détecteur Covid19 est en ligne !\n\n"
Tester une interface API fonctionnelle, telle que **/predict/heatmap ** pour renvoyer le résultat de la classification et de la heatmap d'une image radiographique d'entrée. L'image entrante est codée en based64 avant d'être envoyée via HTTP POST conformément aux définitions de l'API :
%%time# Importation de la bibliothèque des requêtes import argparse import base64import requests# définition d'un point d'entrée ap pour api API_ENDPOINT = "http://172.17.0.1:8051/covid19/api/v1/predict/heatmap"image_path = './Covid_M/all/test/covid/nejmoa2001191_f3-PA.jpeg' #image_path = './Covid_M/all/test/normal/NORMAL2-IM-1400-0001.jpeg' #image_path = './Covid_M/all/test/pneumonia_bac/person1940_bacteria_4859.jpeg' b64_image = "" # Encoding the JPG,PNG,etc. image to base64 format with open(image_path, "rb") as imageFile: b64_image = base64.b64encode(imageFile.read())# données à envoyer à l'api data = {'b64': b64_image}# envoi d'une requête post et enregistrement de la réponse en tant qu'objet réponse r = requests.post(url=API_ENDPOINT, data=data)print(r.status_code, r.text)# extraction de la réponse print("{}".format(r.text))
Toutes ces [images de test ont également été téléchargées sur GitHub] (https://github.com/zhongli1990/Covid19-X-Rays/tree/master/all/test). Le résultat du code ci-dessus sera comme ça:
200 {"Input_Image":"http://localhost:8051/static/source/0198f0ae-85a0-470b-bc31-dc1918c15b9620200906-170443.png","Output_Heatmap":"http://localhost:8051/static/result/Covid19_98_0198f0ae-85a0-470b-bc31-dc1918c15b9620200906-170443.png.png","X-Ray_Classfication_Raw_Result":[[0.805902302,0.15601939,0.038078323]],"X-Ray_Classification_Covid19_Probability":0.98,"X-Ray_Classification_Result":"Covid-19 POSITIVE","model_name":"Customised Incpetion V3"}
{"Input_Image":"http://localhost:8051/static/source/0198f0ae-85a0-470b-bc31-dc1918c15b9620200906-170443.png","Output_Heatmap":"http://localhost:8051/static/result/Covid19_98_0198f0ae-85a0-470b-bc31-dc1918c15b9620200906-170443.png.png","X-Ray_Classfication_Raw_Result":[[0.805902302,0.15601939,0.038078323]],"X-Ray_Classification_Covid19_Probability":0.98,"X-Ray_Classification_Result":"Covid-19 POSITIVE","model_name":"Customised Incpetion V3"}
CPU times: user 16 ms, sys: 0 ns, total: 16 ms
Wall time: 946 ms
3. Les applications de démonstration de services pour les tests benchmarkés
Nous avons mis en place une instance d'équilibreur de charge HAProxy. Nous avons également démarré un service Flask avec 4 travailleurs, et un service FastAPI avec 4 travailleurs également.
Pourquoi ne pas créer 8x processus Pyhon directement dans le fichier Notebook, pour émuler 8x clients API simultanés envoyant des requêtes dans les API du service de démonstration, pour voir ce qui se passe ?
#from concurrent.futures import ThreadPoolExecutor as PoolExecutor from concurrent.futures import ProcessPoolExecutor as PoolExecutor import http.client import socket import timestart = time.time()#laodbalancer: API_ENDPOINT_LB = "http://172.17.0.1:8088/covid19/api/v1/predict/heatmap" API_ENDPOINT_FLASK = "http://172.17.0.1:8052/covid19/api/v1/predict/heatmap" API_ENDPOINT_FastAPI = "http://172.17.0.1:8057/covid19/api/v1/predict/heatmap" def get_it(url): try: # boucle sur les images for imagePathTest in imagePathsTest: b64_image = "" with open(imagePathTest, "rb") as imageFile: b64_image = base64.b64encode(imageFile.read()) data = {'b64': b64_image} r = requests.post(url, data=data) #print(imagePathTest, r.status_code, r.text) return r except socket.timeout: # dans un scénario du monde réel, vous feriez probablement quelque chose si le # socket passe en timeout passurls = [API_ENDPOINT_LB, API_ENDPOINT_LB, API_ENDPOINT_LB, API_ENDPOINT_LB, API_ENDPOINT_LB, API_ENDPOINT_LB, API_ENDPOINT_LB, API_ENDPOINT_LB]with PoolExecutor(max_workers=16) as executor: for _ in executor.map(get_it, urls): pass print("--- %s seconds ---" % (time.time() - start))
Il a donc fallu 74 secondes pour traiter 8x27 = 216 images de test. Cette pile de démonstration à charge équilibrée était capable de traiter 3 images par seconde (en renvoyant les résultats de la classification et de la heatmap aux clients) :
--- 74.37691688537598 seconds ---
À partir de la commande Top de la session Putty, nous pouvons voir que 8x processus de serveur (4x gunicorn + 4 unicorn/python) ont commencé à monter en puissance dès que les scripts de référence ci-dessus ont commencé à être exécutés.
.png)
Suivant
Cet article n'est qu'un point de départ pour mettre en place une pile de déploiement "All-in-Docker AI demo" comme cadre de test. J'espère ensuite ajouter d'autres interfaces de démonstration API, telles que l'interface de prédiction des soins intensifs Covid-19, idéalement conforme à la norme FHIR R4, etc. Cela pourrait également être un banc d'essai pour explorer une intégration plus étroite avec les capacités ML hébergées par IRIS. Au fil du temps, il peut être utilisé comme un cadre de test (et un cadre assez simple) pour intercepter de plus en plus de modèles ML ou DL spécialisés au fur et à mesure que nous avançons sur divers aspects de l'IA, notamment l'imagerie médicale, la santé de la population ou la prédiction personnalisée, le traitement automatique des langues, etc. J'ai également dressé une liste de souhaits à la toute fin de l'article précédent (dans sa section "Next" (Suivant)).